前回の記事では、ランダムフォレストと重回帰分析の予測精度をサンプル数ごとに比較しました。今回は、LightGBMと重回帰分析の比較結果をご紹介します。
特に医療データ分析に携わる方にとって、**「どのモデルを選ぶべきか」**の参考になれば幸いです。
LightGBMとは?
LightGBM(ライト・ジービーエム)は、**勾配ブースティング(Gradient Boosting)**に基づく高速・高精度な機械学習モデルで、Microsoftにより開発されました。
大規模なデータや複雑な非線形パターンを扱う場面で優れた性能を発揮します。
主な特徴
- 非常に高速で、大規模データでも実用的に動作
- 非線形データのような複雑なデータでも高い精度が期待できる
- **SHAP値(Shapley Additive Explanations)**を用いた予測根拠の可視化が可能
- 複数の決定木を段階的に構築し、誤差を補正していくブースティング方式。情報量の多い特徴から分割するリーフワイズ成長によって高い精度を実現
- 医療・社会科学など、解釈性が求められる領域でも活用が進んでいる
ランダムフォレストが「多数の木の平均」で予測するのに対し、LightGBMは「前の木の誤差を次の木で修正」していきます。そのため、効率が良くサンプル数が非常に多い場合や非線形性が強いデータに対して有効です。
「非線形データ?」と思った方はこちらの記事で解説しています。一言で言うと、データ間の関係を直線で表せないデータのことです。
SHAP値とは?
SHAP値(SHapley Additive exPlanations)は、モデルの予測結果に対する各変数の貢献度を示す指標です。医療従事者の方にもイメージしやすいよう簡単に説明すると:
- グラフの上に表示される変数ほど、予測への影響が大きい
- 赤い点は、その変数が高い値のとき予測値を上昇させる傾向を示す
- 青い点は、その変数が高い値のとき予測値を下降させる傾向を示す
例えば、「認知機能スコア(MMSE)が高いほど退院時の機能(FIM)が高い」という関係性が赤い点の分布として視覚的に確認できるため、結果の解釈がしやすくなります。
実験の内容
以下の条件で、重回帰分析と**LightGBM(ハイパーパラメータは初期設定)**を比較しました。
実験条件
- 目的変数:fim_out(退院時の機能スコア)
- 説明変数:fim_in、age、mmse、paralysis
- データ:非線形性を含む機械的に生成したデータ(FIMを模倣)。データの分布は下のグラフのようになっています。
- 評価指標:MSE(平均二乗誤差)…大きな予測誤差に敏感
- サンプル数:50から始め、50ずつ増加させて800まで検証
- 各サンプルサイズで10回ずつモデル構築と評価を行い、MSEの平均と標準偏差を算出
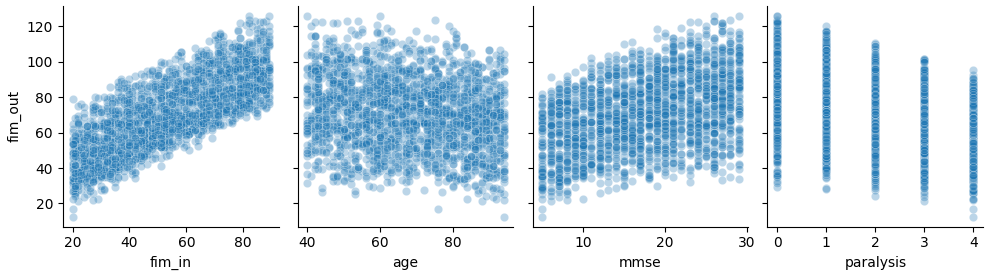
※ちなみに、線形データを使った場合は、ランダムフォレストと重回帰分析の精度にほとんど差が出ません。そのため今回は非線形データを使用しています。
⚠️ 注意:このデータは機械的に生成した架空の物です。実際の患者特性や臨床データを再現したものではありません。
結果と考察
結果の概要
以下のグラフはlightGBMと重回帰分析それぞれで予測したときの誤差を記録したグラフです。実線が誤差の平均値です。それぞれのサンプル数で10回ずつ予測を繰り返したときに、どれぐらい誤差にばらつきが出たかを色つきの帯で示しています。
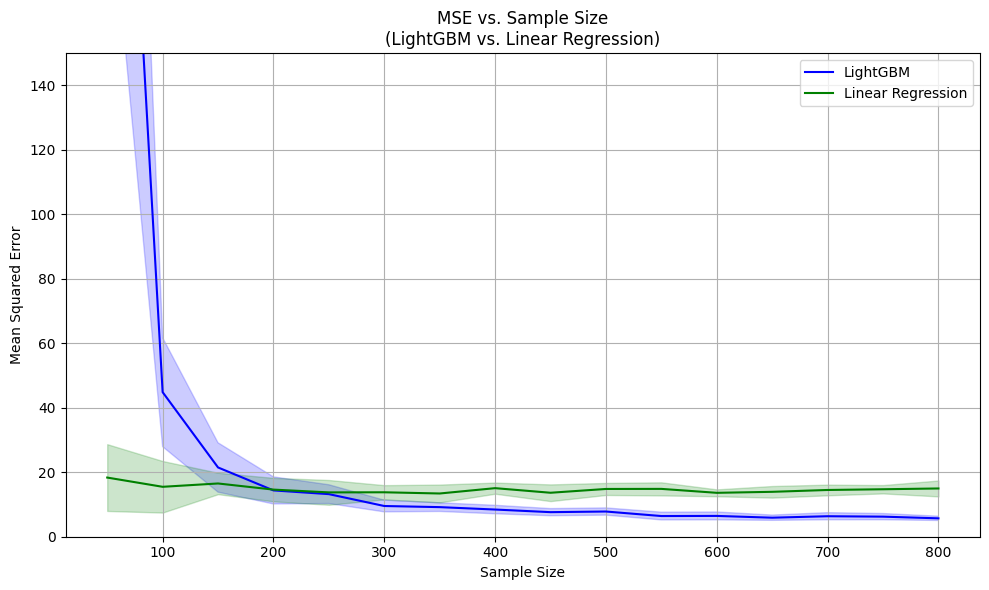
- 150件程度までの少数データでは、LightGBMの精度は低い
- 200件あたりで、重回帰分析との精度の逆転がおこる
- 400件以上では明確な差が現れ、LightGBMが優位に
SHAP値の可視化
以下の二つのグラフはSHAP値をプロットしたものです。上がlightGBMのSHAP値で下が回帰分析のSHAP値です。左右の位置がSHAP値の大きさ、色は説明変数自体の大きさを示しています。fim_inを見てみると、左右の広がりが変数の中で一番大きく、SHAP値が大きいものほど色が赤くなっています。これは変数の中ではfim_inがfim_outへの影響が一番大きく、fim_inが大きいほどfim_outも大きくなることを表しています。
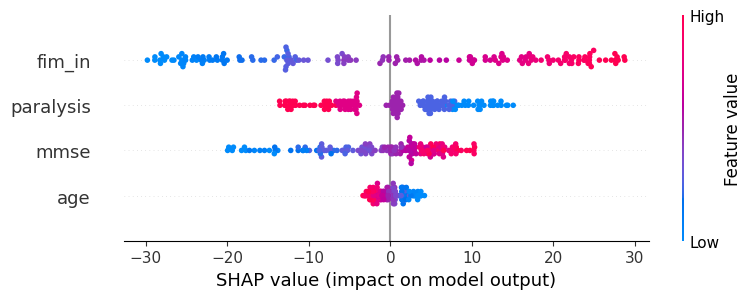
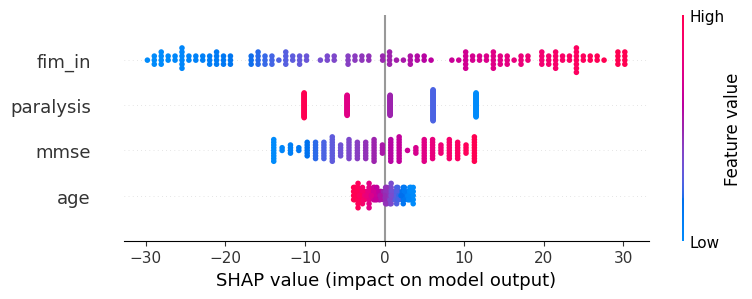
結果からの考察
少数データ(〜150件)
- LightGBMは過学習の傾向が見られ、テスト精度が安定しませんでした。
改善のためのヒント
- 学習率を下げる
- 木の深さ(max_depth)を制限する
- 正則化や最小データ数などのハイパーパラメータを調整
多数データ(400件〜)
- 十分なサンプルがあれば、LightGBMは高精度かつ高速に学習を進められました。以前ディープラーニングでもモデル比較を行いまいしたが、今回のデータではディープラーニングより高速で同等の精度を出せていました。
解釈性について
LightGBMは「ブラックボックス」な印象を持たれがちですが、SHAPによる可視化で予測根拠が明示され、医療現場でも一定の説明責任を果たせるモデルと言えるでしょう。
モデル選択のための比較表
| 特性 | 重回帰分析 | LightGBM |
| 少数データでの精度 | ◎ 安定 | △ 過学習の傾向 |
| 多数データでの精度 | △ 精度頭打ち | ◎ 高精度・高スケーラビリティ |
| 変数同士の関連性の分かりやすさ | ◎ 高い | ○ SHAPで補完可能 |
| 利用の手軽さ | ◎ 単純 | △ 環境構築・知識が必要 |
| 非線形性への対応 | × 対応不可 | ◎ 自然に捉える |
| 計算コスト | 速い | ◎ 精度を考えると非常に速い |
| 外れ値への頑健性 | △ 影響受けやすい | ○ 一定の耐性あり |
実務的な選択ガイド
サンプル数による選択目安
- 〜200件:重回帰分析 → 精度・解釈ともに安定
- 200〜400件:目的に応じて選択(精度を重視するならLightGBM)
- 400件以上:LightGBMが最有力候補
医療現場での活用可能性
- 重回帰分析:学習コストが低くて扱いやすく、従来からの信頼がある。ほとんどの統計ソフトに標準で搭載されている。
- LightGBM:習得にはやや学習コストがかかるが、一度使いこなせば高精度かつ解釈可能なツール
まとめ:使うならどっち?
| 条件 | おすすめモデル |
| データが少ない/結果をすぐ説明したい | 重回帰分析(解釈性◎) |
| データが多い/非線形性が強い | LightGBM(精度・速度◎) |
| 医療現場でもAIを使いたいが信頼性が気になる | SHAP可視化付きLightGBM |
全体的には重回帰分析とランダムフォレストの比較と同じような結果となりました。ただ、計算速度にはランダムフォレストとlightGBMで大きな違いがあり、大規模データではその差が効いてくると思います。
なお、データ分析に詳しい人が周りにいない場合は、重回帰分析の方が手軽でいいかもしれません。
ちなみに、lightGBMは決定木ベースのブースティングモデルの一つです。このようなモデルは他にもあり、CatboostやXGboostなどがあります。こちらの記事ではlightGBM・Catboost・XGboostで精度の評価を行っています。ぜひご覧ください。
実験に使用したコード(参考)
import pandas as pd
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LinearRegression
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import mean_squared_error
import shap
import matplotlib.pyplot as plt
import lightgbm as lgb
# データ生成
np.random.seed(42)
n = 2000
fim_in = np.random.randint(20, 90, n)
age = np.random.randint(40, 95, n)
mmse = np.random.randint(5, 30, n)
paralysis = np.random.randint(0, 5, n)
noise = np.random.normal(0, 5, n)
fim_out = (
fim_in * 3 +
30 * np.sin(mmse / 10) -
(age - 35) / 2 +
(30 - paralysis**2) / 4 +
mmse * (6 - paralysis) +
noise
)
fim_out = fim_out / (fim_out.max() / 126)
df = pd.DataFrame({
'fim_in': fim_in,
'age': age,
'mmse': mmse,
'paralysis': paralysis,
'fim_out': fim_out
})
# 特徴量と目的変数
features = ["fim_in", "age", "mmse", "paralysis"]
target = "fim_out"
sample_sizes = [50, 100, 150, 200, 250, 300, 350, 400, 450, 500, 550, 600, 650, 700, 750, 800]
num_iterations = 10
results_lr = []
results_rf = []
results_std_lr = []
results_std_rf = []
for size in sample_sizes:
mse_list_lr = []
mse_list_rf = []
for i in range(num_iterations):
sample_df = df.sample(n=size, random_state=i)
X = sample_df[features]
y = sample_df[target]
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=i)
# 線形回帰モデル(スケーリングあり)
scaler = StandardScaler()
X_train_scaled = scaler.fit_transform(X_train)
X_test_scaled = scaler.transform(X_test)
model_lr = LinearRegression()
model_lr.fit(X_train_scaled, y_train)
y_pred_lr = model_lr.predict(X_test_scaled)
mse_lr = mean_squared_error(y_test, y_pred_lr)
mse_list_lr.append(mse_lr)
# LightGBMモデル
model_rf = lgb.LGBMRegressor()
model_rf.fit(X_train, y_train)
y_pred_rf = model_rf.predict(X_test)
mse_rf = mean_squared_error(y_test, y_pred_rf)
mse_list_rf.append(mse_rf)
# SHAP解析(1回だけ)
if size == max(sample_sizes) and i == 0:
print("SHAP解析を実行中...")
explainer_rf = shap.Explainer(model_rf, X_train)
shap_values_rf = explainer_rf(X_test)
shap.summary_plot(shap_values_rf, X_test, plot_type="bar")
shap.summary_plot(shap_values_rf, X_test)
plt.title("LightGBM SHAP Feature Importance")
plt.show()
explainer_lr = shap.Explainer(model_lr, X_train_scaled)
shap_values_lr = explainer_lr(X_test_scaled)
shap.summary_plot(shap_values_lr, X_test_scaled, feature_names=features, plot_type="bar")
shap.summary_plot(shap_values_lr, X_test_scaled, feature_names=features)
plt.title("Linear Regression SHAP Feature Importance")
plt.show()
results_lr.append(np.mean(mse_list_lr))
results_rf.append(np.mean(mse_list_rf))
results_std_lr.append(np.std(mse_list_lr))
results_std_rf.append(np.std(mse_list_rf))
# 結果表示
print("サンプルサイズ:", sample_sizes)
print("線形回帰の平均MSE:", results_lr)
print("LightGBMの平均MSE:", results_rf)
# MSE推移の可視化(平均のみ)
plt.plot(sample_sizes, results_lr, label="Linear Regression", marker="o")
plt.plot(sample_sizes, results_rf, label="LightGBM", marker="s")
plt.xlabel("Sample Size")
plt.ylabel("Mean Squared Error")
plt.title("モデル性能の比較 (MSE)")
plt.legend()
plt.grid(True)
plt.show()
# MSEと標準偏差の推移(エラーバー付き)
plt.figure(figsize=(10, 6))
results_lr = np.array(results_lr)
results_rf = np.array(results_rf)
results_std_lr = np.array(results_std_lr)
results_std_rf = np.array(results_std_rf)
plt.plot(sample_sizes, results_rf, label="LightGBM", color="blue")
plt.fill_between(sample_sizes, results_rf - results_std_rf, results_rf + results_std_rf, color="blue", alpha=0.2)
plt.plot(sample_sizes, results_lr, label="Linear Regression", color="green")
plt.fill_between(sample_sizes, results_lr - results_std_lr, results_lr + results_std_lr, color="green", alpha=0.2)
plt.xlabel("Sample Size")
plt.ylabel("Mean Squared Error")
plt.title("MSE vs. Sample Size\n(LightGBM vs. Linear Regression)")
plt.ylim(0,150)
plt.legend()
plt.grid(True)
plt.tight_layout()
plt.show()




コメント